Quantitative und System-Biologie

Unsere Forschungsgruppe nutzt eine Kombination von in silico- (computergestützten) Methoden mit Daten aus in vitro-, ex vivo- und in vivo-Experimenten, um die Signalwege des Proteasoms zur Regulierung des Immunsystems zu untersuchen.

Proteasom-katalysiertes Peptid-Spleißen
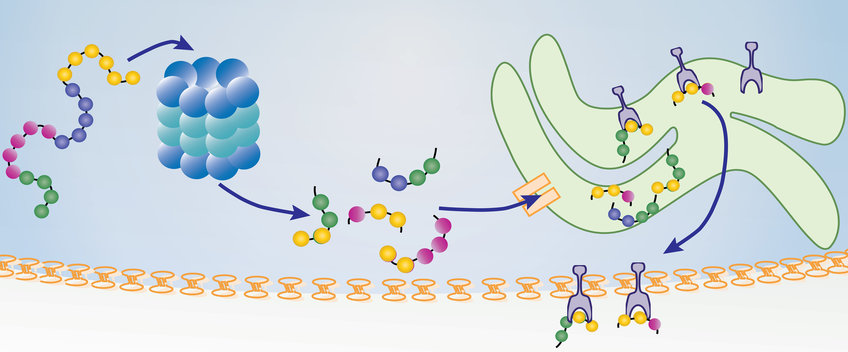
Das Proteasom ist ein Enzymkomplex, der für den Proteinabbau in der Zelle verantwortlich ist. Neben seiner Funktion im Proteinmetabolismus reguliert das Proteasom das menschliche Immunsystem durch Antigenpräsentation, für welche das Proteasom die Mehrheit der an MHC-Klasse-I-bindenden Epitope generiert. Diese Epitope können durch einfaches Schneiden oder durch Schneiden und erneutes Zusammensetzen gebildet werden. Letzterer Prozess wird als Proteasom-katalysiertes Peptid-Spleißen (proteasome-catalyzed peptide splicing) bezeichnet und generiert etwa ein Drittel aller an MHC-Klasse-I gebundenen Epitope.
Woran wir arbeiten
Wir haben diverse mathematische und bioinformatische Methoden entwickelt, um die Details der Peptid-Hydrolyse und des Peptid-Spleißens sowie deren Funktion im MHC-Klasse-I-Signalweg zu untersuchen. Dazu zählen Algorithmen zur Identifizierung von Peptiden, die mithilfe des Proteasoms gespleißt wurden (proteasome-generated spliced peptides, PSPs) basierend auf ex cellulo-massenspektrometrischen Daten sowie Methoden zur Klassifizierung und Charakterisierung der PSPs. In Zukunft werden wir diese Methoden und Erkenntnisse nutzen, um Peptid-Spleißen mit Computerprogrammen vorherzusagen.
Unsere Gruppe hat daher ihren Schwerpunkt in der Entwicklung und Anwendung diverser Methoden, wie zum Beispiel der Entwicklung effizienter massenspektrometrischer Suchalgorithmen, maschinellem Lernen und der Entwicklung von Algorithmen zur dynamischen Modellierung molekularer Prozesse sowie zur Parameterbestimmung.
Therapeutische Relevanz
Das Proteasom ist bereits heute Zielobjekt therapeutischer Ansätze gegen Krebs und Infektionserkrankungen, jedoch muss sein volles Potenzial noch erforscht werden. Die Arbeit unserer Gruppe wird solche translationalen Aspekte unterstützen, wobei insbesondere die Epitop-Identifizierung zur Anwendung in der Immuntherapie und Impfstoffentwicklung gegen Krebs und Infektionskrankheiten im Mittelpunkt steht.





