Zelluläre Biochemie
Struktur und Funktion von Spleißosomen
Die Bildung von Proteinen in Zellen höherer Lebewesen ist ein komplexer, mehrstufiger Vorgang. Zunächst wird die genetische Information für ein Protein von der DNA in eine Arbeitskopie, die Precursor-Boten-RNA (Prä-mRNA), umgeschrieben. Diese RNA Kopien können jedoch erst dann für die Proteinherstellung verwendet werden, wenn durch präzisen Zuschnitt die nicht benötigten internen Bereiche, sogenannte „Introns“, entfernt und die informationsrelevanten Bereiche, „Exons“, neu verknüpft worden sind. Dieser Reifungsprozess wird als mRNA-Spleißen bezeichnet. Erst diese reife mRNA kann aus dem Zellkern ins Zytoplasma transportiert werden und den Ribosomen als Matrize zur Bildung von Proteinen dienen. Diese Exon-Intron-Anordnung bietet dem Organismus große Vorteile: Durch alternatives Spleißen, das heißt durch wahlweises Verknüpfen unterschiedlicher Exons, können von einem Gen mRNAs für verschiedene Proteine hergestellt werden. Alternatives Spleißen stellt damit eine zusätzliche Ebene der Regulation der Genexpression dar, die die genetische Kapazität höherer Eukaryonten enorm erhöht. Dies erklärt auch, warum der Mensch mit etwas mehr als nur 20.000 proteinkodierenden Genen in seinem Genom auskommt.
Die mRNA-Spleißreaktion erfolgt in zwei Schritten über Phosphoester-Transferreaktionen und wird im Zellkern durch die molekulare Maschine des Spleißosoms bewerkstelligt. Spleißosomen setzen sich aus weit über 100 Proteinen und fünf kleinen RNA-Molekülen (den snRNAs U1, U2, U4, U5 und U6) zusammen und sind damit gigantische, sehr proteinreiche Nanomaschinen. Viele dieser Bausteine sind in stabilen Subkomplexen organisiert. So lagern sich z. B. ca. 50 spleißosomale Proteine mit den snRNAs zu RNA-Proteinpartikeln, den snRNPs (small nuclear ribonucleoproteins) U1, U2 und dem U4/U6.U5 tri-snRNP, zusammen.

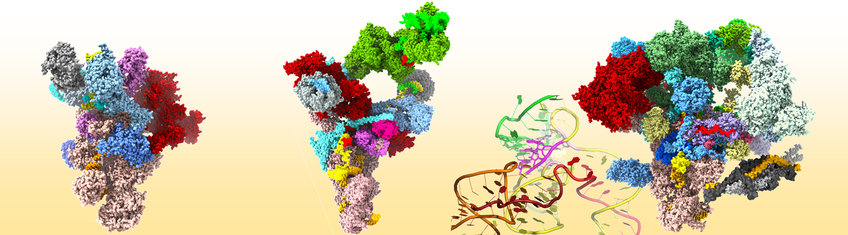
Spleißosomen existieren im Zellkern nicht als vorgefertigte Maschinen, sondern werden auf jedem zu spleißenden Intron jeweils von neuem aus ihren Bausteinen aufgebaut (Abb 1). Zunächst erkennen und binden die snRNPs U1 und U2 zusammen mit einigen Helferproteinen Anfang und Ende eines Introns. Die Bindung des U4/U6.U5 tri-snRNP führt dann zur Ausbildung des sogenannten B Komplexes. Dieser Multi-Mega-Dalton-Komplex besitzt jedoch noch kein aktives katalytisches Zentrum. Schrittweise dramatische Strukturänderungen des B Komplex Spleißosoms verändern die Konformationen der snRNAs und die ursprüngliche biochemische Zusammensetzung des B Komplexes. Dabei bildet sich ein komplexes Netzwerk von RNA-RNA-Wechselwirkungen zwischen der Prä-mRNA und den snRNAs U2, U5 und U6 aus, das als Herzstück des katalytischen Zentrums zu betrachten ist (Abb. 2). Das katalytisch aktivierte Spleißosom ist jetzt in der Lage, den ersten Schritt der Spleißreaktion zu vollziehen, wobei das C Komplex Spleißosom entsteht. Nachdem der zweite katalytische Schritt erfolgt ist, wird das Spleißosom zusammen mit dem herausgeschnittenen Intron von der reifen mRNA abgelöst und in seine Einzelteile dissoziiert. Sowohl snRNAs als auch spleißosomale Proteine sind für die Funktion des Spleißosoms essentiell. So sind sie u. a. an der Erkennung der Spleißstellen und der Ausbildung des katalytischen Zentrums beteiligt. Weiterhin sind eine Reihe energieverbrauchender Enzyme, sogenannte RNA-Helikasen, entscheidend an den schrittweise erfolgenden Umlagerungen des Spleißosoms beteiligt (Abb. 1).

Abb. 2: Dynamik des spleißosomalen RNA-RNA-Netzwerks. Gezeigt sind die wichtigsten RNA-Basenpaarungen im prä-katalytischen B Komplex (linkes Bild) und im aktivierten Spleißosom (rechtes Bild). Für die Aktivierung des Spleißosoms werden die Basenpaarungen zwischen U4- und U6-snRNA sowie zwischen U1-snRNA und der 5‘ Spleißstelle dissoziiert. Dafür geht U6-snRNA mit U2-snRNA und der 5‘ Spleißstelle neue Basenpaarungen ein. Dieses RNA-Netzwerk stellt den Kern des katalytischen Zentrums dar (gelb unterlegt). Die Positionierung der 5‘ Spleißstelle im Exon 1 wird zusätzlich durch Wechselwirkungen der U5-snRNA mit dem 3‘ Ende des Exon 1 unterstützt. Die snRNAs sind cartoonhaft in ihren Sekundärstrukturmodellen gezeigt. Das Intron der pre-mRNA (violett) ist als dünne Linie dargestellt.
Ein wesentliches Ziel unserer Forschung liegt darin, die Funktionsweise und Struktur der Spleißmaschinerie zu verstehen. Dabei steht zum Einen die Frage im Vordergrund, wie die Strukturumwandlungen des Spleißosoms während seines Arbeitszyklus vonstattengehen und wie sie reguliert werden. Zum Anderen wollen wir die Frage klären, wie das katalytische Zentrum des Spleißosoms aufgebaut ist, d. h. besteht es wie bei einem Ribozym nur aus RNA-Bestandteilen, oder agiert es eher wie ein RNP Enzym, in dem RNA und Proteine gleichermaßen zur Katalyse beitragen? Um diese Fragen beantworten zu können, verwenden wir einen integrierten experimentellen Ansatz, der ein breites methodisches Spektrum umfasst. So analysieren wir die Funktion der Proteine und snRNA Moleküle beim Spleißen mit biochemischen und molekulargenetischen Methoden, wobei wir uns vornehmlich auf die Untersuchung von Spleißosomen aus Humanzellen und der Bäckerhefe konzentrieren. Gleichzeitig untersuchen wir mit der Kryo-Elektronenmikroskopie und der Röntgenkristallografie sowie mit massenspektrometrischen und fluoreszenzspektroskopischen Methoden die Raumstruktur und strukturelle Dynamik isolierter Spleißosomen.


